|
1 |
52.4 |
645683.7 |
617.2 |
|
2 |
56.8 |
724813.0 |
625.8 |
|
3 |
48.3 |
685812.4 |
643.9 |
表-2 Blast串行程序的性能数据
(时间单位为ms,已经排除数据库数据驻留内存对第二次测试的影响。)
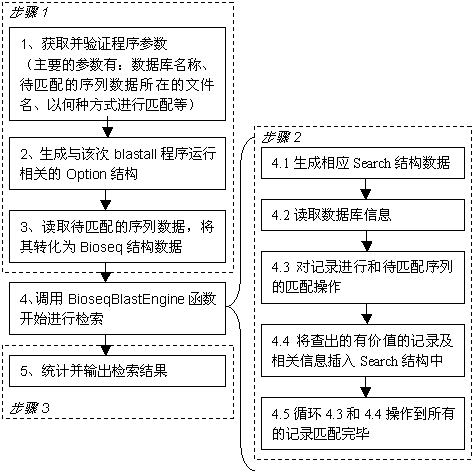
图-1 Blast串行检索程序流程
4.2. Blast的并行化
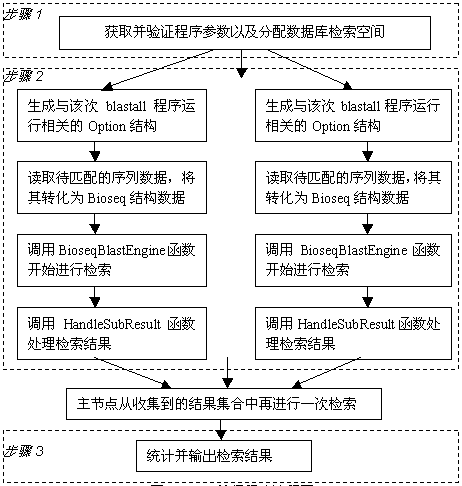
由串行Blast的工作流程可以清楚的发现,Blast在进行检索时采用的方法是循环匹配所有的记录。我们只需将这种循环匹配平均地分配到并行系统的各个节点上,各个节点分别执行各自的匹配操作,最后将匹配的结果统计起来就可以初步实现Blast程序的并行操作。对Blast实行并行化实际上就是将整个检索空间分解成若干个子空间,为各个子节点分配一个子空间,子节点在各自的子空间进行检索,检索完成后,由主控节点归纳统计各个子节点上的结果,然后生成并打印最后的统计结果。
由于Blast程序中牵涉到许多的与生物学相关的计算,检索结果的数据中包含各种在检索过程中生成的数据,有些数据的值和检索的空间范围有关系。如果采取每个子节点生成自己的检索结果的方法,由于每个子节点的结果会由于搜索的集合不同而有所不同,在最后主控节点综合所有结果时,需要归纳所有子节点的结果,这样就干涉了Blast程序的内部计算。同时各个子节点需要向主节点传递检索结果,而检索结果中包含大量的指针。
有一种比较好的并行方法,为各个子节点分配一个记录检索空间,子节点将可能满足条件的记录标记出来,然后传送给主控节点,主控节点再在这个可能的检索空间上进行一次检索,获得检索结果。这种方法只需要将各个子节点检索结果中的记录序号传递给主节点,主节点在搜集到所有子节点上的记录序号后,再在这个记录序号集上进行一次检索,就可以直接在主节点中生成完全正确的检索结果。虽然这种方法由于在子节点完成检索后,主节点还要进行一次检索,在时间上可能会慢一些。但是它大大减少了子节点和主节点之间的通讯量,并且不需要干涉检索结果具体的生成过程,便于程序的并行实现。
Blast程序并行方法的示意图如下:
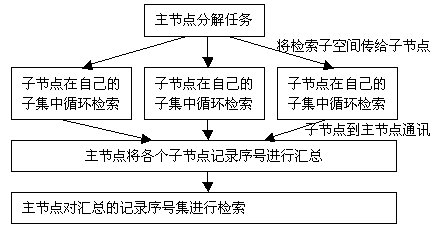
图-2 Blast的并行方法
4.3. Blast并行程序的实现和性能
4.3.1. Blast并行程序的实现
Blast的并行化采用MPI并行开发环境在曙光2000实现,实现过程如下:
1. 由主节点读取数据库信息,根据参与查询节点的数目将数据库的记录分解成与节点匹配的几个查询范围,然后将待查询的序列数据和数据库名称以及查询的记录范围,传递给各个子节点;
2. 修改BioseqBlastEngine函数,在该函数中加入两个参数,用来接受数据库查询的记录范围,各个子节点开始在自己的范围内开始查询,查询完成后生成查询结果——RESEARCH结构数据;
3. 增加处理查询结果的函数,HandleSubResult,这个函数在每个子节点上运行处理各自的查询结果,从结果中读取符合条件的记录序号,并将序列序号传递给主节点;
4. 主节点获取所有子节点的查询结果后,在查询结果的范围中再进行一次查询,筛选出最符合条件的若干记录,并重新生成查询结果——RESEARCH结构数据,并打印该数据。
Blast并行程序的流程如下: