|
有目标性位点的靶向核酸酶被广泛用于基因组编辑。2013 年第一次将 Crispr/cas9 技术用于基因组编辑以来,这个领域发生革命性的变化。作为基因组编辑工具,Crispr/cas9 最成功的地方是它可以轻松的设计向导性 RNA 序列将 Cas9 引导到基因组的特定位点上,然后通过 Cas9 的核酸酶切割活性造成 DNA 断裂。最近的几项研究,成功地应用 Crispr/cas9 纠正动物模型,体细胞和体外诱导的多能干细胞中引起疾病的突变基因,这也对基因组编辑技术应用于临床燃起了希望。我们对近期应用 Crispr/cas9 技术用于基因治疗研究做一综述。
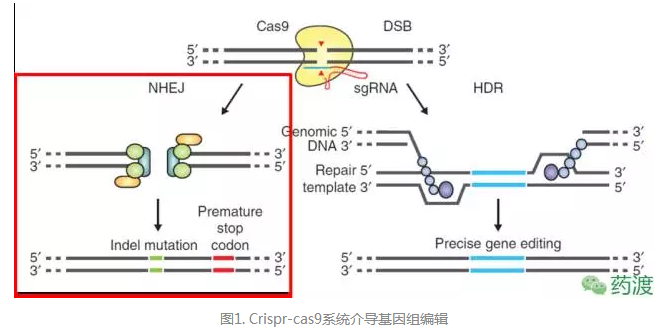
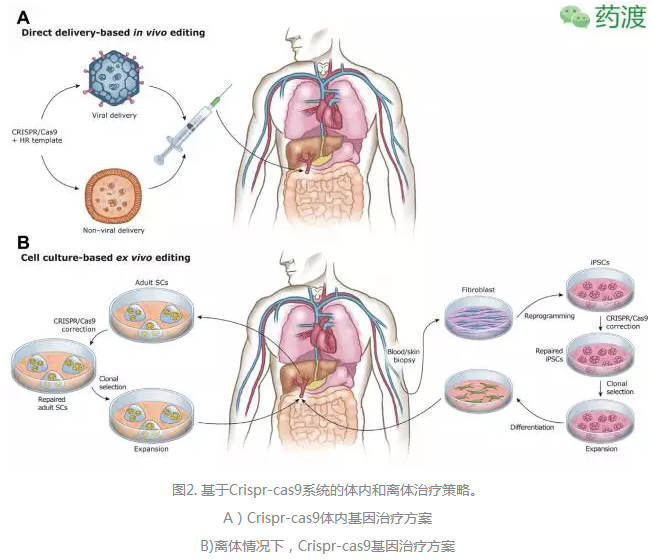
规律成簇间隔短回文重复序列( CRISPRs )配合与它相关的Cas蛋白赋予细菌或古生菌防御外源入侵的核酸,如噬菌体和质粒 DNA[1]。目前为止已经发现的 Crispr/cas9 系统有三种类型,研究得最为透彻的是第二种类型[2]。在细菌防御反应中,外源性DNA会被切割成很多小段,插入到CRISPR位点旁,然后被转录成Cr-RNA前体,继续通过剪切变成成熟的 Cr-RNA 。相应的反义链上会经过转录和转录后加工,形成一个互补的 traCr-RNA ,跟Cas9核酸酶形成核蛋白复合体继续识别和切割入侵的外源 DNA[2]。2012 年,Emmanuelle Charpentier 和 Jennifer Doudna 用二型 Crispr/cas9 技术对酿脓链球菌基因组进行编辑[3]。Cr-RNA 和 traCr-RNA 形成一个向导性的 sgRNA,通过 Watson-Crick 互补配对原则定位到基因组特定位点,招募Cas9核酸酶过来切割 DNA,造成 DNA 双链断裂反应( DSB )[3, 4]。Crispr/Cas9复合体通过两种不同的机制对基因组进行编辑。第一种方式是在缺少同源性DNA模板时,DSB 损伤修复主要是通过非同源末端连接( NHEJ ),这是一种容忍差错的修复,会引起小的插入和缺失。第二种方式是在同源性 DNA 模板存在情况下,通过同源重组的方法进行精确修复,也可以对特定的核苷酸序列进行替换(如图1所示)[5, 6]。 在 Crispr/cas9 之前,主要是用锌指核酸酶和 TALEN 技术进行 DNA 编辑。但是,涉及这种特异性识别DNA序列的蛋白基序非常耗时,这严重影响了技术的发展[7, 8]。Crispr/cas9技术具有锌指核酸酶和TALEN技术无法比拟的可操作性,广泛应用于各种物种的DNA编辑,包括人和灵长动物[9-12]。这些研究表明 Crispr/Cas9 有望应用于基因治疗,可以改变引起疾病的基因的核酸序列。我们对最近应用 Crispr/cas9 技术用于基因治疗研究做一归纳。首先介绍一下,体内条件下,Crispr/cas9 直接针对受精卵和成体动物(如图 2A 所示)。再综述一下,通过体外培养细胞,用 Crispr/Cas9 体外敲除基因,这这些修饰过的细胞回输入病人体内,成功控制病人疾病的案例(如图 2B 所示)。
1.Crispr/cas9对受精卵基因组编辑 共同注射 Cas9 的信使 RNA 或者蛋白,sgRNA 和 HDR 模板到受精卵或早期胚胎中可以改变所有细胞的基因组,包括生殖细胞。因此,这种方法造成的基因组改造可以遗传到子代个体中,为消除家族性的遗传疾病提供可能。上海生化细胞所的李劲松研究员首次报道用 Crispr/cas9 技术修复小鼠胚胎中引起疾病的突变基因。他们矫正了小鼠白内障显性突变基因 Crygc。他们将 Cas9的信使 RNA 和靶向 Crygc 基因的 sgRNA 同时注射到受精卵中,修复时以同源染色体上正常的基因做模板,进行同源重组修复,最后矫正了的小鼠的白内障[13]。另一项研究用 Crispr/Cas9 在小鼠胚胎中矫正肌萎缩蛋白基因,这种疾病是X染色体连锁的遗传性疾病[14]。在这项研究中,科研人员们同时注射Cas9 信使 RNA,sgRNA 和单链寡核苷酸片段一起到受精卵中进行HDR修复。尽管作者们只获得了部分细胞得到矫正的嵌合体,是由于 DNA 编辑发生后受精卵分化的之后,通过自然选择后依然得到了表型完全矫正的小鼠。在最近的一项研究中,研究人员用 Crispr/cas9 技术在人胚胎细胞中中进行了基因组编辑[15],引发了一些生物伦理方面的争论[16, 17]。研究报道说在人胚胎的三原核合子中修改血红素β基因,这个基因突变会造成地中海贫血症。研究人员也发现了 Crispr/cas9 技术存在着脱靶风险,带来很多不知道的 DNA 编辑,现在的技术用于临床还存在风险。现有的产前检测技术,比如体外受精后遗传筛选可以筛选掉存在先生性疾病的胚胎。生殖细胞基因组编辑主要也为那些需要优化孩子一些性状基因,如智商,相貌等等提供了可能。处于更多的安全和伦理问题,并不建议将 Crispr/cas9 技术用于人合子基因组编辑合法化。
2.Crispr/cas9对体细胞的体内编辑 Yin 等人运用 Crispr/cas9 技术成功地对出生后Ⅰ型酪氨酸血症小鼠进行矫正[18]。Ⅰ型酪氨酸血症在遗传学上是由于缺少 fumarylacetoacetate hydrolase ( FAH ) 酶引起的,导致肝细胞代谢毒物的积累而造成细胞死亡。通过尾静脉注射 Cas9 载体,特异性的 sgRNA 和 DNA 模板在小鼠肝脏细胞进行同源重组修复。突变的 FAH 酶被矫正,肝细胞毒性减弱,小鼠的体重回升。酪氨酸血症非常适合用 Crispr/cas9 系统进行基因治疗,起初只有大约 0.4% 的肝细胞被矫正,后来通过肝细胞增殖和突变的肝细胞的死亡,整个肝脏机会恢复正常。 另外一项体内研究是关于敲除小鼠肝脏细胞中 proprotein convertase subtilisin /kexin type 9 ( PCSK9 ) [19]。PCSK9从肝细胞中分泌到血浆中,作为低密度脂蛋白受体激动剂,它控制低密度脂蛋白的摄取与降解。天然出现的PCSK9 突变会减少血液中的胆固醇水平。为了模拟天然出现的情况,研究者用 Crispr/cas9 腺病毒载体在小鼠肝脏细胞中敲除 PCSK9。这导致 PCSK9 蛋白水平的降低和低密度脂蛋白受体的升高。更为重要的是,这种方法是通过NHEJ方法造成肝细胞PCSK9的突变,效率高达50%,这足够用于临床治疗。 Lin等人的研究证明了 Crispr/cas9 介导的基因组编辑同样可以用于乙肝治疗。许多乙肝病人,HBV的长期慢性感染导致肝硬化和肝癌[20]。尽管已经对乙肝病人的抗病毒治疗有很多方法,仍不能彻底消除乙肝病人肝脏中的病毒。这是由于乙肝病毒基因组的复制中间体是一种类似质粒一样的共价闭合环状结构,非常稳定。在这项研究中,从小鼠的尾静脉注射 HBV 表达载体来模拟乙肝病毒感染的情况,同时注射 Crispr/cas9 系统靶向 HBV 表达载体,发现HBV表达载体会被清除。在小鼠血液中乙肝病毒表面抗原浓度会减弱。由于这个小鼠模型还不是产生正常乙肝病毒感染情况下共价闭合环状复制中间体,研究人员又在人的细胞系中感染鸭乙肝病毒,并用 Crispr/cas9 系统靶向病毒基因组,结果进一步证明 Crispr/cas9 的编辑作用完全可以彻底清除乙肝病毒。毫无疑问,任何残留的病毒基因组都会使得病毒死灰复燃,重新感染。因此,发展高效的 Crispr/cas9 系统对临床治疗很多必要。 在前面介绍临床前的验证研究都是在小鼠模型进行了 Crispr/cas9 进行基因治疗。真正用于临床治疗还需要克服几个问题。第一个问题是,提供 Crispr/cas9 系统的效率和安全性。在过去的20多年中,对传统的外源表达载体用于基因治疗研究很多[21, 22]。这些研究研发了很多病毒和非病毒基因治疗载体,这对运送 Crispr/cas9 系统很有帮助。腺相关病毒穿梭载体由于它本身的高效率,适用细胞类型广泛,低细胞毒性和低免疫原性而更具潜力。最新的研究可以把 Cas9和 sgRNA 放到一个腺相关载体中,他们将 Staphylococcus aureus 的小分子量 Cas9 克隆进去[23]。第二个问题是 Crispr/cas9 用于体内基因治疗的话,相对于 NHEJ,HDR 低效率怎么解决?这限制了它应用于消除基因功能和矫正后增加细胞生存优势的基因治疗。解决这个问题的办法是用配对的Cas9切口酶造成 DNA 单链断裂,相对于 NHEJ,这会增加 HDR 修复的比例[24, 25]。第三个问题是 Crispr/cas9 用于疾病治疗的最大的安全问题是它的脱靶效应[26, 27]。这些不需要的双链断裂会造成突变和染色体的重排。Crispr/cas9 脱靶问题还存在争议,最近发明的全基因组分析方法可以控制它的脱靶问题[28-31]。这些分析方法表明 sgRNA 之间的脱靶概率相差很大,从零到 150 个位点。切割效率从 0.03% 到 87%。即使非常低的脱靶效应对于体内基因治疗也是问题,如果是涉及到了肿瘤相关基因。因此,提高 Crispr/cas9 系统特异性尤为重要。两项最近的研究表明在 sgRNA 5’端加上两个 G [32]或者将 sgRNA 缩短到17个核苷酸[33]。用配对的 Cas9 切口酶造成DNA单链断裂而不是引起双链断裂[34, 35],或者使用 dCas9-FokⅠ融合蛋白都可以大大减少脱靶突变效应[36, 37]。 3.Crispr/cas9对体细胞的离体编辑 活体外的基因编辑,需要先培养病人来源干细胞或者前体细胞,经过体外的基因组编辑后再回输入病人体内。2007 年,山中伸弥发明了诱导人成纤维细胞变成多能干细胞[38]。诱导性多能干细胞可以无限增殖并可以分化成任何类型的细胞,这对离体的基因治疗提供了可能。另外,过去的几年中,研究人员建立了体外培养成体干细胞的实验方法[39-41]。这种方法得到的干细胞并没有去分化的步骤,也比诱导性多能干细胞更为安全。 Crispr/cas9 离体的基因编辑的实验性研究起始于用它对诱导性多能干细胞进行编辑,用来治疗β珠蛋白生成障碍性贫血[42]。目前,治疗β珠蛋白生成障碍性贫血的唯一方法是回输组织相容的健康捐献者的造血干细胞。矫正病人来源的诱导性的多能干细胞为生产可移植的造血干细胞提供一个备选方案。研究人员从 β 珠蛋白生成障碍性贫血病人身上取下成纤维细胞,然后诱导成多能干细胞,转染靶向性的 Crispr/cas9 和 DNA 模板进行 HDR 修复,同源重组修复通过抗性基因筛选,筛选后通过转座酶切除,再将这样的多能性干细胞诱导成红细胞的前体细胞,然后用于移植。Hans Clevers 主持的一项研究证明 Crispr/cas9 系统可用于原代的成体干细胞的基因组编辑。在体外培养的小肠类器官模型中,可以无限地扩增多能肠干细胞,然后用 Crispr/cas9 矫正囊肿性纤维化[43]。囊肿性纤维化是一种单基因性遗传病,是由于 CFTR 基因突变后造成胃肠道,肺支气管的粘液积累造成一定的症状,如呼吸困难,反复感染。 分离培养了囊肿性纤维化病人的肠隐窝,在体外三维培养成类器官,然后转染进 靶向 CFTR 基因座的 Crispr/cas9 和 DNA 模板进行同源重组修复。 嘌呤霉素筛选后,通过测序和功能试验证明成功矫正 CFTR 基因。进而又检测了这个实验出现的脱靶效应很少。 两个独立的实验室运用 Crispr/cas9 技术矫正 DMD 病人来源诱导性多能干细胞和永生化细胞系[44, 45]。DMD 是由于抗肌萎缩蛋白基因的突变,引起肌肉纤维结构的改变。失去这个基因的功能会使得肌肉纤维结构的改变,最终引起骨骼肌,呼吸和心肌的衰退。第一个实验室用 Crispr/cas9 联合 DNA 模板通过同源重组的方法恢复抗肌萎缩蛋白全长基因,原先的 DMD 病人的诱导性多能干细胞缺少第44位的外显子。修复的诱导性多能干细胞通过筛选后可诱导分化成表达野生抗肌萎缩蛋白基因的骨骼肌细胞。第二个实验室在 DMD 病人来源的成心肌细胞系中缺失单个或多个外显子恢复抗肌萎缩蛋白的读码框。多重基因编辑可以建立一个缺失突变热点的 45 到 55 位外显子,几乎 60% 的 DMD病人都时基因突变发生在该区域。这种缺失修复后,无论在体外还是在移植到小鼠体内都能回复抗肌萎缩蛋白的表达。 最后,另外一项基于 Crispr/cas9 技术的离体基因编辑研究处理 HIV 感染[46]。HIV感染的周期是先整合到宿主的免疫细胞T细胞基因组中,然后作为模板进行病毒复制。如果基因组转录沉默会导致潜伏感染。由于病毒潜伏在长寿命的记忆性T细胞中,即使存在着抗逆转录病毒的药物,感染会无限地持续。运用基因组编辑技术,研究人员近来用了两种方案对针对 HIV 潜伏感染,病毒基因组被核酸酶靶向后消除整合在T细胞基因组中的HIV病毒 DNA 。基于这项研究,最近在原代艾滋病人的 CD4 阳性T细胞中,用 Crispr/cas9 系统靶向高度保守的 HIV 病毒 TLR 区域后,发现病毒的产生和潜在感染源得到消除。研究人员又进一步证明了,经过靶向 TLR 的 Crispr/cas9 的 HIV 可感染细胞,而这样的细胞是从诱导性多能干细胞分化而来的,可以抵抗新的 HIV 感染。另一个方法是,通过基因组编辑去除赋予 HIV 病毒抵抗性的 CCR5 受体,它是HIV感染T细胞的辅助性受体。Mandal 等人用 Crispr/cas9 编辑技术敲除 CD341 阳性造血干细胞上的 CCR5 [47]。他们用靶向性 CCR5 的 Crispr/cas9 载体转染 CD341 阳性造血干细胞,敲除效率大约为 30%。同时,他们也证明敲除 CCR5 的多能造血干细胞移植到小鼠体内,仍保持分化成多个血液系统细胞的潜能,很少脱靶。更为重要的是,锌指核酸酶靶向的 CCR5 已经成功地在临床上通过试验[48]。 4.展望 离体的基因治疗最大的优势在于可以体外筛选和分析矫正的细胞。因此,只有被矫正而又没有脱靶的细胞才会被选择回输进病人体内。由于有筛选检测的过程,离体情况下的 Crispr/cas9 介导的基因组编辑的效率和精确性比起直接体内操作要求显得宽松一些。缺点在于,离体编辑需要通过体外培养扩增细胞,在培养过程中可能会造成基因组的改变。诱导性多能干细胞在重编程和扩增过程中容易积累突变和发生 CNV [49, 50]。尽管最近的研究表明体外三维培养的成体干细胞遗传稳定性很高,但是体外细胞扩增仍然存在安全问题。离体基因治疗的另外一个问题是高效的移植正确的细胞。现在已经在临床上有很成熟造血干细胞移植的技术,但是其它组织,如肝和肌肉细胞移植技术还在改进中。毫无疑问,尽管存在这么多问题,这么多障碍, Crispr/cas9 技术仍然是临床基因治疗的巨大期望,未来会有翻天覆地的变化。 参考文献 1. Wiedenheft, B., S.H. Sternberg, andJ.A. Doudna, RNA-guided genetic silencingsystems in bacteria and archaea. Nature, 2012. 482(7385): p. 331-338. 2. Doudna, J.A. and E. Charpentier, The new frontier of genome engineering withCRISPR-Cas9. Science, 2014. 346(6213):p. 1258096. 3. Jinek, M., et al., A programmable dual-RNA–guided DNAendonuclease in adaptive bacterial immunity. Science, 2012. 337(6096): p. 816-821. 4. Mali, P., et al., RNA-guided human genome engineering via Cas9. Science, 2013. 339(6121): p. 823-826. 5. Barnes, D.E., Non-homologous end joining as a mechanism of DNA repair. Currentbiology, 2001. 11(12): p. R455-R457. 6. Bosch, M.v.d., P.H. Lohman, and A.Pastink, DNA double-strand break repairby homologous recombination. Biological chemistry, 2002. 383(6): p. 873-892. 7. Joung, J.K. and J.D. Sander, TALENs: a widely applicable technology fortargeted genome editing. Nature reviews Molecular cell biology, 2013. 14(1): p. 49-55. 8. Urnov, F.D., et al., Genome editing with engineered zinc fingernucleases. Nature Reviews Genetics, 2010. 11(9): p. 636-646. 9. Cho, S.W., et al., Targeted genome engineering in human cellswith the Cas9 RNA-guided endonuclease. Nature biotechnology, 2013. 31(3): p. 230-232. 10. Cong, L., et al., Multiplex genome engineering using CRISPR/Cas systems. Science,2013. 339(6121): p. 819-823. 11. Wang, H., et al., One-step generation of mice carrying mutations in multiple genes byCRISPR/Cas-mediated genome engineering. Cell, 2013. 153(4): p. 910-918. 12. Niu, Y., et al., Generation of gene-modified cynomolgus monkey via Cas9/RNA-mediatedgene targeting in one-cell embryos. Cell, 2014. 156(4): p. 836-843. 13. Wu, Y., et al., Correction of a genetic disease in mouse via use of CRISPR-Cas9.Cell stem cell, 2013. 13(6): p.659-662. 14. Long, C., et al., Prevention of muscular dystrophy in mice by CRISPR/Cas9–mediatedediting of germline DNA. Science, 2014. 345(6201): p. 1184-1188. 15. Liang, P., et al., CRISPR/Cas9-mediated gene editing in human tripronuclear zygotes.Protein & cell, 2015. 6: p.363-372. 16. Cyranoski, D., Ethics of embryo editing divides scientists. Nature, 2015. 519(7543): p. 272-272. 17. Lanphier, E., et al., Don't edit the human germ line. Nature,2015. 519(7544): p. 410. 18. Yin, H., et al., Genome editing with Cas9 in adult mice corrects a disease mutation andphenotype. Nature biotechnology, 2014. 32(6):p. 551. 19. Ding, Q., et al., Permanent alteration of PCSK9 with in vivo CRISPR-Cas9 genome editing.Circulation research, 2014. 115(5):p. 488-492. 20. Lin, S.-R., et al., The CRISPR/Cas9 system facilitates clearance of the intrahepatic HBVtemplates in vivo. Molecular Therapy—Nucleic Acids, 2014. 3(8): p. e186. 21. Mingozzi, F. and K.A. High, Therapeutic in vivo gene transfer forgenetic disease using AAV: progress and challenges. Nature reviewsgenetics, 2011. 12(5): p. 341-355. 22. Kay, M.A., State-of-the-art gene-based therapies: the road ahead. NatureReviews Genetics, 2011. 12(5): p.316-328. 23. Ran, F.A., et al., In vivo genome editing using Staphylococcus aureus Cas9. Nature,2015. 520(7546): p. 186-191. 24. Ran, F.A., et al., Double nicking by RNA-guided CRISPR Cas9 for enhanced genome editingspecificity. Cell, 2013. 154(6):p. 1380-1389. 25. Osborn, M.J., et al., Fanconi anemia gene editing by theCRISPR/Cas9 system. Human gene therapy, 2014. 26(2): p. 114-126. 26. Pattanayak, V., et al., High-throughput profiling of off-target DNAcleavage reveals RNA-programmed Cas9 nuclease specificity. Naturebiotechnology, 2013. 31(9): p.839-843. 27. Fu, Y., et al., High-frequency off-target mutagenesis induced by CRISPR-Cas nucleasesin human cells. Nature biotechnology, 2013. 31(9): p. 822-826. 28. Frock, R.L., et al., Genome-wide detection of DNA double-strandedbreaks induced by engineered nucleases. Nature biotechnology, 2015. 33(2): p. 179-186. 29. Tsai, S.Q., et al., GUIDE-seq enables genome-wide profiling of off-target cleavage byCRISPR-Cas nucleases. Nature biotechnology, 2015. 33(2): p. 187-197. 30. Kim, D., et al., Digenome-seq: genome-wide profiling of CRISPR-Cas9 off-target effectsin human cells. Nature methods, 2015. 12(3):p. 237-243. 31. Crosetto, N., et al., Nucleotide-resolution DNA double-strandbreak mapping by next-generation sequencing. Nature methods, 2013. 10(4): p. 361-365. 32. Cho, S.W., et al., Analysis of off-target effects of CRISPR/Cas-derived RNA-guidedendonucleases and nickases. Genome research, 2014. 24(1): p. 132-141. 33. Fu, Y., et al., Improving CRISPR-Cas nuclease specificity using truncated guide RNAs.Nature biotechnology, 2014. 32(3):p. 279-284. 34. Kim, E., et al., Precision genome engineering with programmable DNA-nicking enzymes.Genome research, 2012. 22(7): p.1327-1333. 35. Mali, P., et al., CAS9 transcriptional activators for target specificity screening andpaired nickases for cooperative genome engineering. Nature biotechnology,2013. 31(9): p. 833-838. 36. Tsai, S.Q., et al., Dimeric CRISPR RNA-guided FokI nucleases for highly specific genomeediting. Nature biotechnology, 2014. 32(6):p. 569-576. 37. Guilinger, J.P., D.B. Thompson, and D.R.Liu, Fusion of catalytically inactiveCas9 to FokI nuclease improves the specificity of genome modification.Nature biotechnology, 2014. 32(6):p. 577. 38. Takahashi, K., et al., Induction of pluripotent stem cells fromadult human fibroblasts by defined factors. cell, 2007. 131(5): p. 861-872. 39. Huch, M., et al., Long-term culture of genome-stable bipotent stem cells from adult humanliver. Cell, 2015. 160(1): p.299-312. 40. Csaszar, E., et al., Rapid expansion of human hematopoietic stemcells by automated control of inhibitory feedback signaling. Cell stemcell, 2012. 10(2): p. 218-229. 41. Sato, T., et al., Long-term expansion of epithelial organoids from human colon, adenoma,adenocarcinoma, and Barrett's epithelium. Gastroenterology, 2011. 141(5): p. 1762-1772. 42. Xie, F., et al., Seamless gene correction of β-thalassemia mutations in patient-specificiPSCs using CRISPR/Cas9 and piggyBac. Genome research, 2014. 24(9): p. 1526-1533. 43. Schwank, G., et al., Functional repair of CFTR by CRISPR/Cas9 inintestinal stem cell organoids of cystic fibrosis patients. Cell stem cell,2013. 13(6): p. 653-658. 44. Li, H.L., et al., Precise correction of the dystrophin gene in duchenne musculardystrophy patient induced pluripotent stem cells by TALEN and CRISPR-Cas9.Stem cell reports, 2015. 4(1): p.143-154. 45. Ousterout, D.G., et al., Multiplex CRISPR/Cas9-based genome editingfor correction of dystrophin mutations that cause Duchenne muscular dystrophy.Nature communications, 2015. 6. 46. Liao, H.-K., et al., Use of the CRISPR/Cas9 system as anintracellular defense against HIV-1 infection in human cells. Naturecommunications, 2015. 6. 47. Mandal, P.K., et al., Efficient ablation of genes in humanhematopoietic stem and effector cells using CRISPR/Cas9. Cell stem cell,2014. 15(5): p. 643-652. 48. Tebas, P., et al., Gene editing of CCR5 in autologous CD4 T cells of persons infected withHIV. New England Journal of Medicine, 2014. 370(10): p. 901-910. 49. Ji, J., et al., Elevated coding mutation rate during the reprogramming of human somaticcells into induced pluripotent stem cells. Stem cells, 2012. 30(3): p. 435-440. 50. Gore, A., et al., Somatic coding mutations in human induced pluripotent stem cells.Nature, 2011. 471(7336): p. 63-67. (责任编辑:泉水) |
【综述】治疗性CRISPR/cas9技术研究进展
时间:2017-01-04 15:49来源:药渡 作者:未知
顶一下
(0)
0%
踩一下
(0)
0%
------分隔线----------------------------
- 上一篇:纳米热疗软化肿瘤 改善治疗
- 下一篇:北大第一医院牟向东:临床医学要有创新思维
- 发表评论
-
- 最新评论 进入详细评论页>>
- 推荐内容
-
- 小胶质细胞在人类大脑发育中的关
研究人员通过将小胶质细胞(大脑免疫细胞)整合到实验室培养...
- 呼吸影响瞳孔大小
摘要 研究人员发现,呼吸会影响瞳孔大小,吸气时瞳孔最小,呼...
- 为什么有些人能生动回忆梦境,而
摘要 一项新研究探讨了为什么有些人能生动回忆梦境,而有些人...
- Science Advance:光催化激活神经Ca2+信号
近日,杭师大材化学院刘俊秋教授团队/生科院沈万华教授团队携...
- 体生长抑素信号在大脑中的作用
作者:宾夕法尼亚州立大学 Sam Sholtis 新研究表明,由大脑中许多...
- 协和发现国内首例“阴性艾滋病患
HIV抗体检测为常见的艾滋病筛查手段,阳性一般意味着感染,阴...
- 小胶质细胞在人类大脑发育中的关