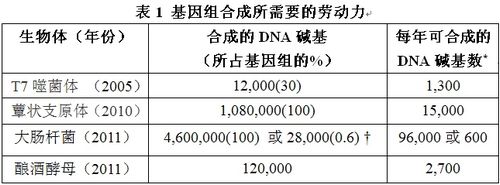
科学家们开始对酵母的基因组进行大规模修饰,以使其更加稳定、更具可操作性和可演变性。值得注意的是,这个一部分天然、一部分合成的酵母细胞的生理功能和繁殖性能都没有明显的不良反应。 酿酒酵母(Saccharomyces cerevisiae)是一种模式生物,因此,它是地球上人们了解得最全面的生物系统之一。但是,它内部功能的复杂性仍然让生物工程学家如同在迷雾中,并且这种复杂性继续为实验研究提供源源不断的研究素材。如果科学家能够“重构”模式生物——也就是说,对它的基因组进行重新编码,使其更简单、更适宜人类了解以及对其修补——那么,建立在这些生物体之上的科学技术和生物技术就能够得到不断发展。2011年9月22日出版的《自然》杂志的471页,Dymond 等人发表了为实现这个目标的一个重大进展:构建功能性和部分人工合成的酿酒酵母的基因组。 一段时间以来,重写基因组以满足人类研究的需要是合成生物学的既定目标。但是,基因组重新编码所需要耗费的劳动力和费用,再加上对这种改变的不确定性,意味着对基因组重编程只能进行少量的尝试。之前值得注意的例子包括:病毒基因组12,000个碱基对(大约30%)的重构,以及大肠杆菌“琥珀”终止密码子(翻译为终止信号的核苷酸序列)的移除,这项技术使得研究人员能够随意地部分重编码细菌遗传密码子。据此,美国马里兰州罗科维尔的奎格文特研究所(J. Craig Venter Institute)的研究人员已经从头合成了一个支原体的基因组,并将它注入到了一个活细胞中。 Dymond等人的研究更进了一步,他们开始研究真核生物(例如真菌、植物和动物),真核生物拥有比细菌更庞大且更为复杂的基因组。更具体地说,作者现在已经利用人工合成的DNA取代了酿酒酵母其中两条染色体中的一部分——染色体IX末端的90,000个碱基和染色体VI末端的30,000个碱基。他们的最终目标是,利用人工合成的序列替代具有1200万个碱基对的整个基因组。 为了插入人工合成的DNA,Dymond等人完全移除了天然酵母染色体中的20个区域。这些区域的绝大部分都是重复序列或者是非功能性和冗余的序列。同时,作者重编码每个基因时都多加了一个长500个碱基的“水印”——这个序列可以很方便地利用标准实验室方法区分天然序列中的合成DNA,但是不会改变基因编码蛋白质的序列。正如之前有关大肠杆菌的报道,Dymond等人修改了酿酒酵母DNA中的“琥珀”终止密码子,这样就可以在未来对其进行重编码,例如编码非天然氨基酸插入到酵母蛋白质中去。 令人吃惊的是,作者发现,含有经修饰的基因组的酵母细胞与野生型菌株相比,没有明显的生长缺陷,并且在基因表达方面的差异也很小。人为添加的整个DNA序列能够准确地在活细胞中复制,这是对人类工程稳健性的实际证明,或者表示创造者的力量比上帝的指纹更为强大。 除了上面提到的变化,Dymond等人在合成DNA中的每个非必需基因后面以及其它位置引入了一个称为loxPsym位点的序列元素。由于重组酶Cre的存在,这些loxPsym位点能够随机地进行相互重组。因此,作者能够建立一个庞大的酵母基因组库,包含各种结构的基因。我们可以对这个基因库进行筛选,然后找出更适合在给定环境中生活的新的酵母菌株。因为酵母是用来生产酒精、蛋白质和高价值的有机化合物的,所以通过这种方式产生的新菌株可能最终证明是能用于工业生产的,同样地,简化的大肠杆菌已经被证明是生产大量蛋白质的一个很好的平台。 Dymond等人的往后的工作就是要重建整个酵母基因组。然而,鉴于目前完成的合成序列大约只占整个基因组的1%,重建其余的基因仍是一项艰巨的任务。此外,有一个问题是,虽然DNA合成中使用的材料、设备和耗材的总成本一直在稳步下降,但是,整个基因组的构建依然是代价不菲。 更大的问题是劳动力成本。通过对基因合成和修改所消耗的劳动力的比较可以看出(表1),像酵母基因组这样规模的生物体的DNA合成不仅需要大量的科学家还需要新的技术。Dymond具有里程碑意义的工作证实,对自动化的DNA合成和组装技术的需求越来越迫切,基因组的全部合成很可能会取代基因组修饰所用到的零散的技术方法。随着技术进步的不断推进,设计基因组的时代也就近了。
*数据是根据以下公式计算:合成的全部碱基数/(作者人数×3),估计每个人花费在基因合成项目上的平均时间是3年。†这个数据要看到底是整个基因组参与计算,还是仅合成的寡核苷酸参与计算决定。 原文检索: |
酿酒酵母基因组研究新进展
时间:2011-10-21 16:21来源:生命奥妙 作者:
顶一下
(1)
100%
踩一下
(0)
0%
------分隔线----------------------------
- 发表评论
-
- 最新评论 进入详细评论页>>
- 推荐内容
-
- 基因编辑新工具“出炉”以RNA为目
美国科学家日前发现一种新的基于CRISPR系统的基因编辑工具,它以...
- 章鱼和乌贼是RNA编辑高手,但DNA却
任何作家都会在编辑修改故事时抱怨,但改动一个词的后果通常...
- 新疆野苹果再生及CRISPR/Cas9基因编辑
2021-07-30 新疆生态与地理研究所 【字体:大 中 小】 语音播报...
- 美国将开展首例CRISPR抗癌人体试验
据英国《自然》杂志网站、美国《麻省理工技术评论》网站近日...
- 基因编辑技术有望治疗镰状细胞病
一个美国研究小组使用CRISPR-Cas9基因编辑技术,成功修复了镰状...
- DIY基因编辑技术让你无所不能
随着越来越多的新闻媒体报道CRISPR/Cas基因编辑的应用——尤其是...
- 基因编辑新工具“出炉”以RNA为目